Desenmascarando la identidad de las cianobacterias toxigénicas en embalses
6 June, 2019
Investigadores de la UAM y CEDEX desarrollan un nuevo método para identificar cianobacterias tóxicas en embalses. aguasresiduales.info. 04/06/19. Investigadores de la Universidad Autónoma de Madrid (UAM) y el Centro de Estudios y Experimentación de Obras Públicas (CEDEX) han desarrollado un método basado en secuenciación genética masiva para la detección y seguimiento de cianobacterias tóxicas en embalses.
Folleto de cianobacterias
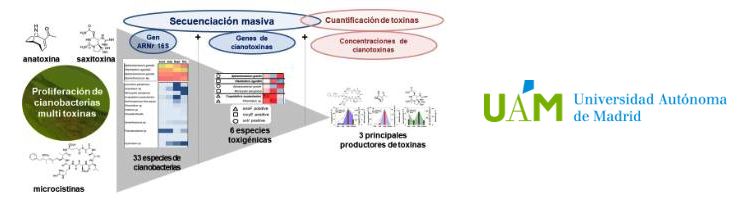 El método combina secuenciación genética masiva y cuantificación de cianotoxinasLos resultados, publicados en Science of The Total Environment, resultan importantes dado que los afloramientos de cianobacterias tóxicas son un problema extendido en los embalses por toda Europa La presencia masiva de cianobacterias en embalses, lo que se conoce como blooms o afloramientos, es un tema de investigación en auge dado su impacto negativo no solo en la biodiversidad acuática sino también en la salud humana, ya que afecta a múltiples usos del agua como abastecimiento, baño, deportes acuáticos o riego. Se trata de un problema global, dado que la presencia de blooms se ha documentado en más de 100 países a día de hoy. En efecto, existen más de 50 géneros de cianobacterias capaces de producir diversas toxinas (cianotoxinas) que afectan directamente la piel (dermatotoxinas), el hígado (hepatotoxinas) o el sistema nervioso (neurotoxinas). Para una debida gestión del agua resulta de vital importancia por tanto identificar correctamente las cianobacterias que habitan los embalses de toda la geografía de la Península Ibérica y su potencial toxicidad, y poder desarrollar así las herramientas de control adecuadas que aseguren una calidad del agua adecuada para su uso y consumo. El desarrollo de una nueva aproximación se probó con éxito en el embalse de Rosarito (Toledo) durante un bloom de cianobacterias en el verano del 2013. Previamente, en este embalse ya se habían detectado tres de las cuatro cianotoxinas más extendidas en Europa: dos neurotoxinas (anatoxina y saxitoxina) y una hepatotoxina (microcistina). Ahora, investigadores de la Universidad Autónoma de Madrid (UAM) y el Centro de Estudios y Experimentación de Obras Públicas (CEDEX), han aplicado un novedoso procedimiento que combina, por un lado, técnicas de secuenciación genética masiva, y por otro, la cuantificación de las toxinas mediante inmunodetección para conocer qué cianobacterias crecieron en aquel bloom y cuáles de ellas fueron las responsables de la toxicidad del agua. Secuenciación masiva combinada Tradicionalmente los estudios sobre cianobacterias en embalses se han abordado mediante el trabajo con cultivos de laboratorio y la observación al microscopio de muestras de agua. Estas metodologías, aunque eficaces, tienen múltiples limitaciones ya que el aislamiento de cultivos de cianobacterias es una tarea tediosa que no refleja muy bien la biodiversidad de bacterias que componen el medio natural, dado que no todas son cultivables en el laboratorio. La identificación directa al microscopio, a su vez, no permite diferenciar con certeza las cepas tóxicas dado que éstas a menudo presentan un aspecto o morfología idéntica a las cepas no tóxicas. En busca de una alternativa más rápida y resolutiva, los investigadores decidieron utilizar una de las denominadas técnicas de secuenciación masiva. Para ello realizaron un análisis en el que combinaron secuencias del gen del ARN ribosómico 16S (un marcador universal de bacterias que permite identificar con mucha precisión incluso aquellas especies en muy baja proporción) con secuencias de cuatro genes involucrados en la producción de los tipos de cianotoxinas más extendidas en Europa para discriminar las cepas tóxicas de aquellas que no lo son. Gracias a este nuevo método, publicado en la revista Science of The Total Environment, el equipo encontró una diversidad de bacterias hasta cuatro veces mayor que la detectada con métodos tradicionales y seis especies de cianobacterias distintas capaces de producir cianotoxinas. Curiosamente, de estas seis especies potencialmente tóxicas sólo dos coincidieron con las cianobacterias más predominantes durante el verano, mientras que las otras cuatro eran especies en una proporción muy baja, de menos del 1%, que habrían sido imposibles de detectar e identificar con las técnicas convencionales de microscopía y cultivos en laboratorio. Además, el tratamiento estadístico de estos datos, cruzado con la cuantificación de toxinas mediante técnicas de detección por anticuerpos (ELISA) permitió ir todavía un paso más allá y precisar que de esas 33 especies detectadas inicialmente eran principalmente tres las que más estaban contribuyendo a las concentraciones de toxinas que se midieron en el embalse. “Este tipo de metodologías facilitaría la prevención y gestión de estos blooms tóxicos mediante la elaboración de bases de datos genéticos de especies tóxicas en cada embalse para de este modo establecer prioridades a nivel de intervención y métodos de seguimiento selectivo de las poblaciones de cianobacterias”, aseguran los autores. “En definitiva, se trataría de poner al servicio de la sociedad en la gestión de aguas una tecnología – la secuenciación masiva- que ya se utiliza en otros ámbitos de interés social como es la biomedicina”, concluyen. _____________________ Referencia bibliográfica Casero, M. C., Velázquez, D., Medina-Cobo, M., Quesada, A., & Cirés, S. (2019). Unmasking the identity of toxigenic cyanobacteria driving a multi-toxin bloom by high-throughput sequencing of cyanotoxins genes and 16S rRNA metabarcoding. Science of The Total Environment, 665, 367-378. https://doi.org/10.1016/j.scitotenv.2019.02.083 Fuente www.uam.es
El método combina secuenciación genética masiva y cuantificación de cianotoxinasLos resultados, publicados en Science of The Total Environment, resultan importantes dado que los afloramientos de cianobacterias tóxicas son un problema extendido en los embalses por toda Europa La presencia masiva de cianobacterias en embalses, lo que se conoce como blooms o afloramientos, es un tema de investigación en auge dado su impacto negativo no solo en la biodiversidad acuática sino también en la salud humana, ya que afecta a múltiples usos del agua como abastecimiento, baño, deportes acuáticos o riego. Se trata de un problema global, dado que la presencia de blooms se ha documentado en más de 100 países a día de hoy. En efecto, existen más de 50 géneros de cianobacterias capaces de producir diversas toxinas (cianotoxinas) que afectan directamente la piel (dermatotoxinas), el hígado (hepatotoxinas) o el sistema nervioso (neurotoxinas). Para una debida gestión del agua resulta de vital importancia por tanto identificar correctamente las cianobacterias que habitan los embalses de toda la geografía de la Península Ibérica y su potencial toxicidad, y poder desarrollar así las herramientas de control adecuadas que aseguren una calidad del agua adecuada para su uso y consumo. El desarrollo de una nueva aproximación se probó con éxito en el embalse de Rosarito (Toledo) durante un bloom de cianobacterias en el verano del 2013. Previamente, en este embalse ya se habían detectado tres de las cuatro cianotoxinas más extendidas en Europa: dos neurotoxinas (anatoxina y saxitoxina) y una hepatotoxina (microcistina). Ahora, investigadores de la Universidad Autónoma de Madrid (UAM) y el Centro de Estudios y Experimentación de Obras Públicas (CEDEX), han aplicado un novedoso procedimiento que combina, por un lado, técnicas de secuenciación genética masiva, y por otro, la cuantificación de las toxinas mediante inmunodetección para conocer qué cianobacterias crecieron en aquel bloom y cuáles de ellas fueron las responsables de la toxicidad del agua. Secuenciación masiva combinada Tradicionalmente los estudios sobre cianobacterias en embalses se han abordado mediante el trabajo con cultivos de laboratorio y la observación al microscopio de muestras de agua. Estas metodologías, aunque eficaces, tienen múltiples limitaciones ya que el aislamiento de cultivos de cianobacterias es una tarea tediosa que no refleja muy bien la biodiversidad de bacterias que componen el medio natural, dado que no todas son cultivables en el laboratorio. La identificación directa al microscopio, a su vez, no permite diferenciar con certeza las cepas tóxicas dado que éstas a menudo presentan un aspecto o morfología idéntica a las cepas no tóxicas. En busca de una alternativa más rápida y resolutiva, los investigadores decidieron utilizar una de las denominadas técnicas de secuenciación masiva. Para ello realizaron un análisis en el que combinaron secuencias del gen del ARN ribosómico 16S (un marcador universal de bacterias que permite identificar con mucha precisión incluso aquellas especies en muy baja proporción) con secuencias de cuatro genes involucrados en la producción de los tipos de cianotoxinas más extendidas en Europa para discriminar las cepas tóxicas de aquellas que no lo son. Gracias a este nuevo método, publicado en la revista Science of The Total Environment, el equipo encontró una diversidad de bacterias hasta cuatro veces mayor que la detectada con métodos tradicionales y seis especies de cianobacterias distintas capaces de producir cianotoxinas. Curiosamente, de estas seis especies potencialmente tóxicas sólo dos coincidieron con las cianobacterias más predominantes durante el verano, mientras que las otras cuatro eran especies en una proporción muy baja, de menos del 1%, que habrían sido imposibles de detectar e identificar con las técnicas convencionales de microscopía y cultivos en laboratorio. Además, el tratamiento estadístico de estos datos, cruzado con la cuantificación de toxinas mediante técnicas de detección por anticuerpos (ELISA) permitió ir todavía un paso más allá y precisar que de esas 33 especies detectadas inicialmente eran principalmente tres las que más estaban contribuyendo a las concentraciones de toxinas que se midieron en el embalse. “Este tipo de metodologías facilitaría la prevención y gestión de estos blooms tóxicos mediante la elaboración de bases de datos genéticos de especies tóxicas en cada embalse para de este modo establecer prioridades a nivel de intervención y métodos de seguimiento selectivo de las poblaciones de cianobacterias”, aseguran los autores. “En definitiva, se trataría de poner al servicio de la sociedad en la gestión de aguas una tecnología – la secuenciación masiva- que ya se utiliza en otros ámbitos de interés social como es la biomedicina”, concluyen. _____________________ Referencia bibliográfica Casero, M. C., Velázquez, D., Medina-Cobo, M., Quesada, A., & Cirés, S. (2019). Unmasking the identity of toxigenic cyanobacteria driving a multi-toxin bloom by high-throughput sequencing of cyanotoxins genes and 16S rRNA metabarcoding. Science of The Total Environment, 665, 367-378. https://doi.org/10.1016/j.scitotenv.2019.02.083 Fuente www.uam.es